grado di avvolgersi alla fine della stringa.
I ricercatori ora sono d’accordo che il 2-point crossover è generalmente migliore del one point.
Tecniche di Crossover
Il GA tradizionale, come è stato già descritto prima, usa il
one point crossover quando i due cromosomi che si accoppiano sono
entrambi tagliati in punti corrispondenti e la sezione dopo i
tagli è cambiata. Comunque, sono stati inventati molti diversi
algoritmi di crossover, che spesso coinvolgono più di un punto
di taglio
DeJong ha studiato l’efficienza del crossover multipoint ed
è arrivato alla conclusione che il due-punti crossover dà un
miglioramento ma che aggiungere più punti crossover riduce
le prestazioni del GA. Il problema con l’aggiunta di
più punti crossover è che i building blocks sono più facili da
spezzare. Comunque un vantaggio di avere molti punti crossover è
che nello spazio del problema si può fare una ricerca più
accurata.
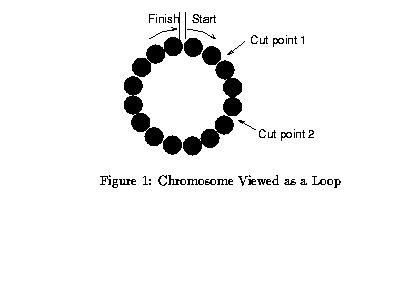
2-point Crossover
In questa tecnica (e in generale nel multi-point) piuttosto che stringhe lineari i cromosomi possono essere considerati come circoli formati dall’unione degli estremi insieme. Per cambiare un segmento da un circolo con un altro proveniente da un altro ciclo, si richiede la selezione di due punti crossover, come mostrato in figura 1. In questa figura il one point crossover può essere visto come un 2-poit crossover, con uno dei punti di taglio fissato all’inizio della stringa. Quindi il 2-point opera come il one-point (cioè cambiando un solo segmento), ma è più generale. Un cromosoma, considerato come un circolo, può contenere più building blocks, poiché sono in

grado di avvolgersi alla fine della stringa.
I ricercatori ora sono d’accordo che il 2-point crossover è
generalmente migliore del one point.
Crossover Uniforme
Questa tecnica completamente differente dal one-point
crossover. Ciascun gene nei figli è creato tramite una copia del
corrispondente gene da uno dei due genitori, scelto in accordo a
una “maschera di crossover” creata in maniera
casuale.
Dove c’è un 1 nella maschera, il gene è copiato dal primo
genitore, e dove c’è uno zero, il gene è copiato dal
secondo genitore, come mostrato in fig.2.
Il processo è ripetuto con i genitori scambiati per produrre un
secondo figlio. Una nuova maschera crossover è generata
casualmente per ciascuna coppia di genitori. Il figlio quindi
contiene una mistura di geni provenienti da ciascun genitore. Il
numero degli effettivi punti crossover non è fissato, ma supererà
L/2, dove L è la lunghezza del cromosoma.

Quale tecnica è la migliore?
Il dibattito su quale sia la migliore tecnica di crossover è
ancora in corso. Syswerda arguisce in favore dell’uniforme.
Riguardo il crossover uniforme gli schemi che hanno un
particolare ordine (l’ordine di uno schema è il numero di
valori dei bit specificati) hanno la stessa probabilità di
essere distrutti, a prescindere dalla lunghezza definita (il
numero di bit tra il primo e l’ultimo bit specificato).
Con il 2-point è la lunghezza definita dello schema che
determina la sua predisposizione alla distruzione, non il suo
ordine. Questo significa che riguardo il crossover uniforme gli
schemi con lunghezze definite corte hanno maggiori
probabilità di essere distrutti, mentre le più lunghe sono
distrutte meno facilmente.
Syswerda inoltre dice che il numero totale delle distruzioni
degli schemi è comunque alta.
Il crossover uniforme ha il vantaggio che l’ordinamento dei
geni è del tutto irrilevante e questo significa che gli
operatori di riordinamento come l’inversione (vedi seguito)
non sono necessari, e noi non dobbiamo preoccuparci di
posizionare i geni per migliorare i buiding blocks.
L’efficienza del GA che usa il 2-point, cade drammaticamente,
se non sono rispettate le raccomandazioni della “building
block hypothesis”. Il crossover uniforme d’altra parte,
continua a lavorare bene almeno tanto bene come un 2-point che
usato con un cromosoma ordinato correttamente. Comunque l’uniforme
sembra essere la tecnica più robusta. Eshelman fornisce una
comparazione estesa di differenti operatori di crossover, incluso
1-2-point, multi-point e uniforme. Questi sono analizzati
teoricamente in termini di deviazione di posizione e
distribuzione, e empiricamente su alcuni problemi. Nessuno
prevale sugli altri e infatti c’erano non più di circa 20%
di differenza nella velocità delle tecniche (forse noi non
dobbiamo preoccuparci molto di quale sia il metodo migliore).
Loro hanno trovato che l’8-point crossover era migliore per
i problemi che avevano provato.
Spears e DeJong sono molto critici riguardo il multi-point e l’uniform
crossover, mentre sono d’accordo con le analisi teoriche che
mostrano che 1 e 2 point crossover sono ottimi. Loro dicono che
il crossover due punti lavorerà male quando la popolazione
è ampiamente convergente, e ciò dovuto alla ridotta
produttività del crossover. Questa è l’abilità dell’operatore
crossover di produrre nuovi cromosomi che nello spazio di ricerca
campionano punti differenti. Quando due cromosomi sono simili, i
segmenti scambiati dal 2-point crossover è probabile che siano
identici, e portano a figli che sono identici ai genitori. Questo
è meno facile che succeda con l’uniform crossover. Loro
descrivono un nuovo operatore 2-point crossover che se produce
due figli identici, sceglie due nuovi cross-point.
Questo operatore era stato cercato per lavorare meglio del
crossover uniforme in un problema test (ma solo leggermente
meglio).
In una pubblicazione successiva hanno concluso che il 2-point
modificato è migliore per grandi popolazioni, ma che la maggiore
distruzione del crossover uniforme è benefica se la dimensione
della popolazione è piccola (in confronto con la complessità
del problema), e quindi da una performance più robusta.
Altre tecniche di Crossover
Molte altre tecniche sono state suggerite. L’idea che il
crossover dovesse essere più forte in certe posizioni sulla
stringa piuttosto che in altre ha qualche fondamento in natura, e
alcuni di questi metodi che sono stati descritti. Il
principio generale è che il GA impara adattativamente quali siti
dovrebbero essere favoriti per il crossover. Questa informazione
è registrata in una stringa punteggiatura, che è essa stessa
parte del cromosoma, e quindi viene incrociata e passata ai
discendenti. In questo modo le stringhe punteggiatura che vanno
in direzione della migliore discendenza saranno esse stesse
propagate attraverso la popolazione.
Goldberg descrive un operatore crossover abbastanza diverso che
si chiama Partially Matched Crossover (PMX), per l’uso in
problemi basati sull’ordine. (In un problema basato sull’ordine
come il problema del commesso viaggiatore, i valori dei geni sono
fissati, e il fitness dipende dall’ordine col quale loro
appaiono). Nel PMX non sono incrociati i valori dei geni, ma
l’ordine con cui appaiono. I figli hanno geni che ereditano
ordinando informazioni da ciascun genitore. Questo elimina
la generazione di figli che violano i vincoli del problema.
Syswerda e Davis descrivono altri operatori basati sull’ordine.